前些天發現了一個巨牛的人工智能學習電子書,通俗易懂,風趣幽默,無廣告,忍不住分享一下給大家。(點擊跳轉人工智能學習資料)
說明:若需要數據附件及python源文件請移步微信公眾號“創享日記”,聯系作者有償獲取!
一、題目
一、題目
根據附件cell.jipg,使用opencv庫或者PIL庫計算細胞核與細胞質的面積比。
二、題目分析
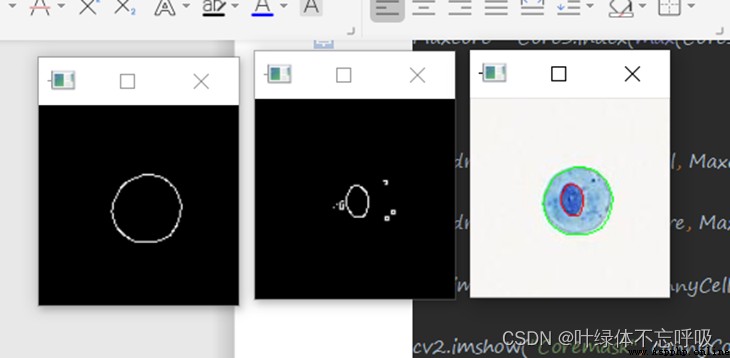
這道題目識別細胞的時候,要分別識別細胞核和細胞膜。對此進行操作的時候,首先需要將圖像轉化成灰度圖像。接著需要將灰度圖像二值化,通過threshold方法傳入灰度圖像和阙值,我們根據細胞核和細胞膜的顏色得到核的阙值為131,細胞質的阙值為220,然後第三個參數傳入最大阙值255,第四個參數為THRESH_BINARY,表示二進制化阙值。
然後根據內核的大小,拋棄掉邊界附近的像素。kernel =np.ones((2,2),np.uint8),在這裡我們使用2*2的內核,它包含了所有的1。之後通過開運算傳入圖像,內核等進行計算。接著通過canny函數進行邊界檢測,查找出輪廓然後提取邊框。通過findContours傳入檢測到的邊緣。最後通過兩個列表儲存邊框面積,獲取最大面積對應的索引框,通過計算得出的細胞核和細胞質的大小相除就得到了核質比
三、代碼
import cv2
import numpy as np
img = cv2.imread("cell.jpg")
# 轉為灰度圖像
gray = cv2.cvtColor(img, cv2.COLOR_BGR2GRAY)
cv2.imshow("gary", gray)
# 二值化
thresh1, Cellmask = cv2.threshold(gray, 220, 255, cv2.THRESH_BINARY)
thresh2, Coremask = cv2.threshold(gray, 131, 255, cv2.THRESH_BINARY)
# 開運算
kernel = np.ones((2, 2), np.uint8)
Cellmask = cv2.morphologyEx(Cellmask, cv2.MORPH_OPEN, kernel)
Coremask = cv2.morphologyEx(Coremask, cv2.MORPH_OPEN, kernel)
# 邊緣檢測
cannyCell = cv2.Canny(Cellmask, 40, 200)
cannyCore = cv2.Canny(Coremask, 40, 200)
# 邊框提取
Cell, Celldata = cv2.findContours(cannyCell, mode=cv2.RETR_EXTERNAL, method=cv2.CHAIN_APPROX_NONE)
Core, Coredata = cv2.findContours(cannyCore, mode=cv2.RETR_EXTERNAL, method=cv2.CHAIN_APPROX_NONE)
# 存儲邊框面積
CellS = []
CoreS = []
for i in range(len(Cell)):
CellS.append(cv2.contourArea(Cell[i]))
for i in range(len(Core)):
CoreS.append(cv2.contourArea(Core[i]))
# 獲取最大面積對應邊框索引
Maxcell = CellS.index(max(CellS))
Maxcore = CoreS.index(max(CoreS))
cv2.drawContours(img, Cell, Maxcell, (0, 255, 0), 1)
cv2.drawContours(img, Core, Maxcore, (0, 0, 255), 1)
cv2.imshow("Cellmask", cannyCell)
cv2.imshow("Coremask", cannyCore)
cv2.imshow("img", img)
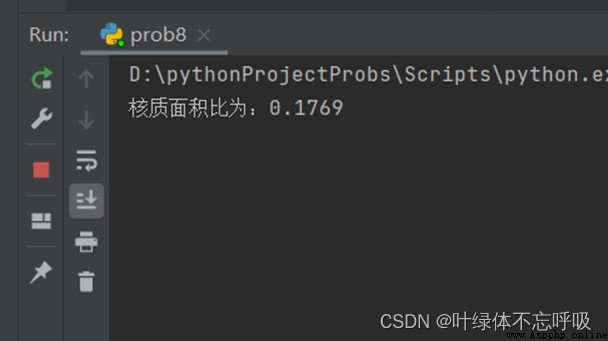
print("核質面積比為:{:.3f}".format(max(CoreS) / (max(CellS)-max(CoreS))))
cv2.waitKey()
四、實驗結果