Kmeans可視化
https://www.naftaliharris.com/blog/visualizing-k-means-clustering/
K-means 有一個著名的解釋:牧師—村民模型:
有四個牧師去郊區布道,一開始牧師們隨意選了幾個布道點,並且把這幾個布道點的情況公告給了郊區所有的村民,於是每個村民到離自己家最近的布道點去聽課。
聽課之後,大家覺得距離太遠了,於是每個牧師統計了一下自己的課上所有的村民的地址,搬到了所有地址的中心地帶,並且在海報上更新了自己的布道點的位置。
牧師每一次移動不可能離所有人都更近,有的人發現A牧師移動以後自己還不如去B牧師處聽課更近,於是每個村民就去了離自己最近的布道點……
就這樣,牧師每個禮拜更新自己的位置,村民根據自己的情況選擇布道點,最終穩定了下來
Kmeans,一種無監督聚類算法,也是最為經典的基於劃分的聚類方法,思想是: 對於給定的樣本集,按照樣本之間的距離大小,將樣本集劃分為K個類。讓類內的點盡量緊密的連在一起,而讓類間的距離盡量的大
步驟:
例子: https://www.bilibili.com/video/BV1py4y1r7DN?share_source=copy_web
有以下6個點,將A3和A4作為初始質心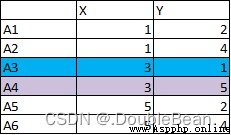
1.計算每個點到質心距離,將距離近的點歸為一類。從下圖可以看到A1與A3的距離是2.24,與A4的距離是3.61,所以A1被歸為A3這一類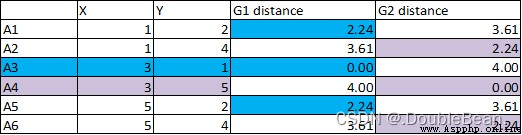
2.將藍色每個點,和紫色每個點的x,y值分別求平均。獲得新的質心
3.計算每個點到質心的新距離,將距離近的點歸為一類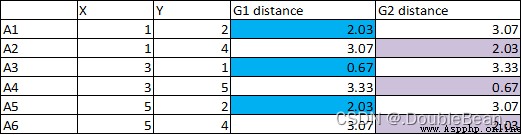
4.由於關聯點沒有變化,所以之後的計算結果不會改變,停止計算
5.藍色類: A1, A3, A5。紫色類:A2, A4, A6。
# 不調庫實現
class my_Kmeans:
def __init__(self, k):
self.k = k
# 距離度量函數: 計算任意點之間的距離
def calc_distance(self, vec1, vec2):
return np.sqrt(np.sum(np.power(vec1 - vec2, 2)))
def fit(self, data):
numSamples, dim = data.shape # numSamples指樣本總數,dim指特征維度
# 隨機並且不重復地選取k個數據作為初始聚類中心點
self.centers_idx = np.random.choice(numSamples, self.k, replace=False) # 得到的是質心點的索引
self.centers = data[self.centers_idx].astype(np.float32) # 初始化聚類中心
# ClusterAssment記錄每個樣本的信息: 第一列記錄樣本所屬聚類中心的索引,第2列記錄樣本與聚類中心的距離的平方(用於SSE指標)
ClusterAssment = np.zeros((numSamples, 2))
ClusterChanged = True
while ClusterChanged:
ClusterChanged = False
# 遍歷所有點求每個點與各個聚類中心點的距離
for i in range(numSamples):
mindist = self.calc_distance(data[i], self.centers[0])
label = 0
for j in range(1, self.k):
distance = self.calc_distance(data[i], self.centers[j])
# 把第i個數據分配到距離最近的聚類中心
if distance < mindist:
mindist = distance
label = j
# print("mindist = {},label = {}".format(mindist, label))
# 判斷樣本所屬聚類中心是否發生了變化,若發生了變化則更新數據
if ClusterAssment[i, 0] != label:
ClusterChanged = True
ClusterAssment[i, :] = label, mindist ** 2
# print(ClusterAssment, '\n')
# 對每個類別的樣本重新求聚類中心,並更新聚類中心
for j in range(self.k):
# 找到所有屬於類中心j的樣本數據
pointsInCluster = data[ClusterAssment[:, 0] == j]
# print("{}: {}".format(j, pointsInCluster))
self.centers[j, :] = (np.mean(pointsInCluster, axis=0).tolist()) # 更新聚類中心的位置
def predict(self, data):
numSamples, dim = data.shape
ClusterAssment = np.zeros((numSamples, 2))
for i in range(numSamples):
mindist = self.calc_distance(data[i], self.centers[0])
label_pred = 0
for j in range(1, self.k):
distance = self.calc_distance(data[i], self.centers[j])
if distance < mindist:
mindist = distance
label_pred = j
ClusterAssment[i, :] = label_pred, mindist ** 2
return self.centers, ClusterAssment
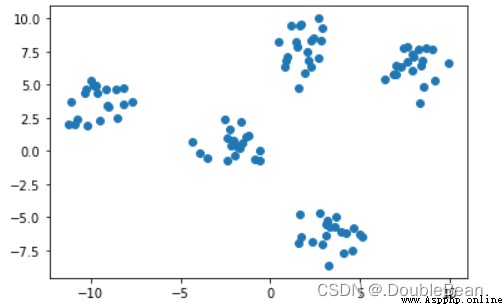
生成一些數據集,來檢驗Kmeans聚類效果
import matplotlib.pyplot as plt
import numpy as np
from sklearn.datasets import make_blobs
data, true_labels = make_blobs(centers = 5, random_state = 20, cluster_std = 1) # data是坐標集合,ture_labels是正確標簽
plt.scatter(data[:, 0], data[:, 1])
plt.show()
# 不調庫實現
model = my_Kmeans(k=5)
model.fit(data)
''' model.fit()函數返回值 centers: 聚類中心坐標 ClusterAssment: 每個樣本的信息: 第一列記錄樣本所屬聚類中心的索引,第2列記錄樣本與聚類中心的距離的平方(用於SSE指標) '''
centers, ClusterAssment = model.predict(data)
plt.figure(figsize=(6, 5))
plt.scatter(data[:, 0], data[:, 1], c = ClusterAssment[:, 0])
plt.scatter(centers[:, 0], centers[:, 1], marker = '*', color = 'r', s = 100) # 繪制類中心點
plt.show()
下圖便是代碼跑出來的結果,多運行幾次會發現不調庫實現的Kmeans聚類結果會各種各樣,即不調庫實現的Kmeans結果大部分情況下都是局部最優,並非全局最優
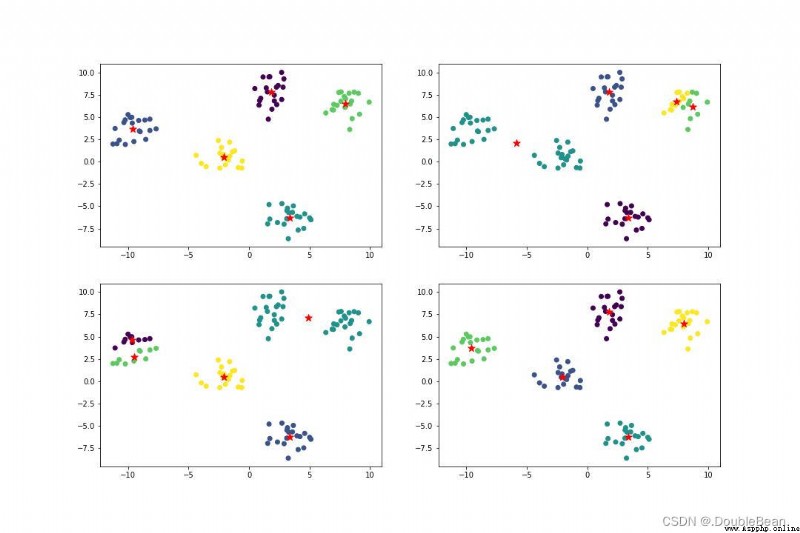
調庫實現的話,無論運行多少次,結果都很好
# 調庫實現
from sklearn.cluster import KMeans
model_1 = KMeans(n_clusters=5)
label_pred_1 = model_1.fit_predict(data)
plt.figure(figsize=(6, 5))
plt.scatter(data[:, 0], data[:, 1], c = label_pred_1)
plt.show()
缺點:
優點:
前面我們提到過K-means存在一些問題,其中一個問題就是K-means對初始的類中心非常敏感,Kmeans++就是專門對初始值的選擇進行優化的
問題點: 隨機選取k個數據,若數據非常靠近,會導致收斂很慢,甚至收斂到局部最小值
可見聚類的結果高度依賴質心的初始化,所以為了解決初始時如何選取k個類中心點的問題,引入了K-means++算法,它的核心思想是,距離已選中心點越遠,被選為下一個中心點的概率越大
那麼問題來了,那是不是選擇距離所有已選中心點最遠的那個點作為下一個中心點呢?並不是,如果這樣做很容易選到異常的離群點,從而影響最終的聚類效果
在講Kmeans之前,先講一下輪盤賭法,基本上很多文章談到輪盤賭算法,都會和遺傳算法,蟻群算法一起解釋
步驟:
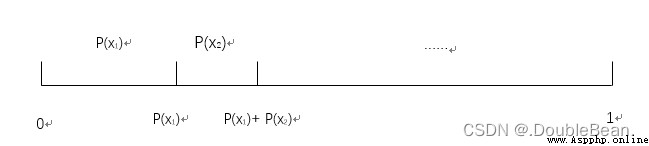
舉例
初始種群適應度為 [169, 576, 64, 361],那麼每個個體被遺傳到下一代群體的概率為:
P ( x 1 ) = f ( x 1 ) ∑ j = 1 m f ( x j ) = 169 169 + 576 + 64 + 361 = 0.14 P ( x 2 ) = f ( x 2 ) ∑ j = 1 m f ( x j ) = 576 169 + 576 + 64 + 361 = 0.49 P ( x 3 ) = f ( x 3 ) ∑ j = 1 m f ( x j ) = 64 169 + 576 + 64 + 361 = 0.06 P ( x 4 ) = f ( x 4 ) ∑ j = 1 m f ( x j ) = 361 169 + 576 + 64 + 361 = 0.31 P(x_1) = \frac{f(x_1)}{\sum_{j=1}^{m} f(x_j)} = \frac{169}{169 + 576 + 64 + 361} = 0.14 \\ P(x_2) = \frac{f(x_2)}{\sum_{j=1}^{m} f(x_j)} = \frac{576}{169 + 576 + 64 + 361} = 0.49 \\ P(x_3) = \frac{f(x_3)}{\sum_{j=1}^{m} f(x_j)} = \frac{64}{169 + 576 + 64 + 361} = 0.06 \\ P(x_4) = \frac{f(x_4)}{\sum_{j=1}^{m} f(x_j)} = \frac{361}{169 + 576 + 64 + 361} = 0.31 P(x1)=∑j=1mf(xj)f(x1)=169+576+64+361169=0.14P(x2)=∑j=1mf(xj)f(x2)=169+576+64+361576=0.49P(x3)=∑j=1mf(xj)f(x3)=169+576+64+36164=0.06P(x4)=∑j=1mf(xj)f(x4)=169+576+64+361361=0.31
每個個體的累計概率為:
q ( x 1 ) = ∑ j = 1 1 P ( x j ) = 0.14 q ( x 2 ) = ∑ j = 1 2 P ( x j ) = 0.14 + 0.49 = 0.63 q ( x 3 ) = ∑ j = 1 3 P ( x j ) = 0.14 + 0.49 + 0.06 = 0.69 q ( x 4 ) = ∑ j = 1 1 P ( x j ) = 0.14 + 0.9 + 0.06 + 0.31 = 1 q(x_1) = \sum_{j=1}^{1} P(x_j) = 0.14 \\ q(x_2) = \sum_{j=1}^{2} P(x_j) = 0.14 + 0.49 = 0.63 \\ q(x_3) = \sum_{j=1}^{3} P(x_j) = 0.14 + 0.49 + 0.06 = 0.69 \\ q(x_4) = \sum_{j=1}^{1} P(x_j) = 0.14 + 0.9 + 0.06 + 0.31 = 1 q(x1)=j=1∑1P(xj)=0.14q(x2)=j=1∑2P(xj)=0.14+0.49=0.63q(x3)=j=1∑3P(xj)=0.14+0.49+0.06=0.69q(x4)=j=1∑1P(xj)=0.14+0.9+0.06+0.31=1
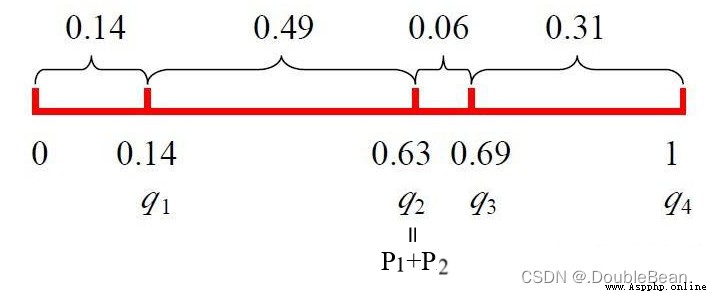
隨機產生一個數r=0.672452,發現是落在[q2, q3]之間,則x3被選中
步驟:
代碼
class my_Kmeans_plus:
def __init__(self, k, init):
self.k = k
self.init = init # init='random'就是標准Kmeans,init='Kmeans++'就是Kmeans++
# 距離度量函數: 計算任意點之間的距離
def calc_distance(self, vec1, vec2):
return np.sqrt(np.sum(np.power(vec1 - vec2, 2)))
''' 利用輪盤法選擇下一個聚類中心 參數: P是各樣本點被選為下一個聚類中心的概率集合 r是區間[0, 1]之間隨機生成的一個數,判斷該數落在哪個區間,則這個區間被選中 返回的是被選中的樣本索引 '''
def RWS(self, P, r):
q = 0 # 累計概率
for i in range(len(P)):
q += P[i] # P[i]表示第i個樣本被選中的概率
if r <= q: # 產生的隨機數在該區間內
return i
def fit(self, data):
numSamples, dim = data.shape # numSamples指樣本總數,dim指特征維度
if self.init == 'random':
# 隨機並且不重復地選取k個數據作為初始聚類中心點
self.centers_idx = np.random.choice(numSamples, self.k, replace = False) # 得到的是質心點的索引
# 默認類別是從0到k-1
self.centers = data[self.centers_idx].astype(np.float32) # 初始化聚類中心
# print(self.centers)
elif self.init == 'Kmeans++':
first = np.random.choice(numSamples) # 隨機選出一個樣本點作為第一個聚類的中心
index_select = [first] # 將聚類中心索引存儲到一個列表中
D = np.zeros((numSamples, 1)) # 初始化D(x)
# 繼續選取k-1個點
for j in range(1, self.k):
for i in range(numSamples):
D[i] = float('inf')
for idx in index_select:
distance = self.calc_distance(data[i], data[idx])
D[i] = min(D[i], distance)
# 計算概率
P = np.square(D) / np.sum(np.square(D))
r = np.random.random() # r為0到1的隨機數
choiced_index = self.RWS(P, r)
index_select.append(choiced_index) # 利用輪盤法選擇下一個聚類中心
# print(index_select)
self.centers = data[index_select].astype(np.float32)
# print(self.centers)
# ClusterAssment記錄每個樣本的信息: 第一列記錄樣本所屬聚類中心的索引,第2列記錄樣本與聚類中心的距離
ClusterAssment = np.zeros((numSamples, 2))
ClusterChanged = True
while ClusterChanged:
ClusterChanged = False
# 遍歷所有點求每個點與各個聚類中心點的距離
for i in range(numSamples):
mindist = self.calc_distance(data[i], self.centers[0])
label = 0
for j in range(1, self.k):
distance = self.calc_distance(data[i], self.centers[j])
# 把第i個數據分配到距離最近的聚類中心
if distance < mindist:
mindist = distance
label = j
# print("mindist = {},label = {}".format(mindist, label))
# 判斷樣本所屬聚類中心是否發生了變化,若發生了變化則更新數據
if ClusterAssment[i, 0] != label:
ClusterChanged = True
ClusterAssment[i, :] = label, mindist
# print(ClusterAssment, '\n')
# 對每個類別的樣本重新求聚類中心,並更新聚類中心
for j in range(self.k):
# 找到所有屬於類中心j的樣本數據
pointsInCluster = data[ClusterAssment[:, 0] == j]
# print("{}: {}".format(j, pointsInCluster))
self.centers[j, :] = (np.mean(pointsInCluster, axis=0).tolist()) # 更新聚類中心的位置
def predict(self, data):
numSamples, dim = data.shape
ClusterAssment = np.zeros((numSamples, 2))
for i in range(numSamples):
mindist = self.calc_distance(data[i], self.centers[0])
label_pred = 0
for j in range(1, self.k):
distance = self.calc_distance(data[i], self.centers[j])
if distance < mindist:
mindist = distance
label_pred = j
ClusterAssment[i, :] = label_pred, mindist ** 2
return self.centers, ClusterAssment
調用自己寫的類
# 不調庫實現Kmeans++
model = my_Kmeans_plus(k = 5, init = 'Kmeans++')
model.fit(data)
centers, ClusterAssment = model.predict(data)
''' # 不調庫實現Kmeans model = my_Kmeans_plus(k = 5, init = 'random') model.fit(data) centers, ClusterAssment = model.predict(data) '''
plt.figure(figsize=(6, 5))
plt.scatter(data[:, 0], data[:, 1], c = ClusterAssment[:, 0])
plt.scatter(centers[:, 0], centers[:, 1], marker = '*', color = 'r', s = 100) # 繪制類中心點
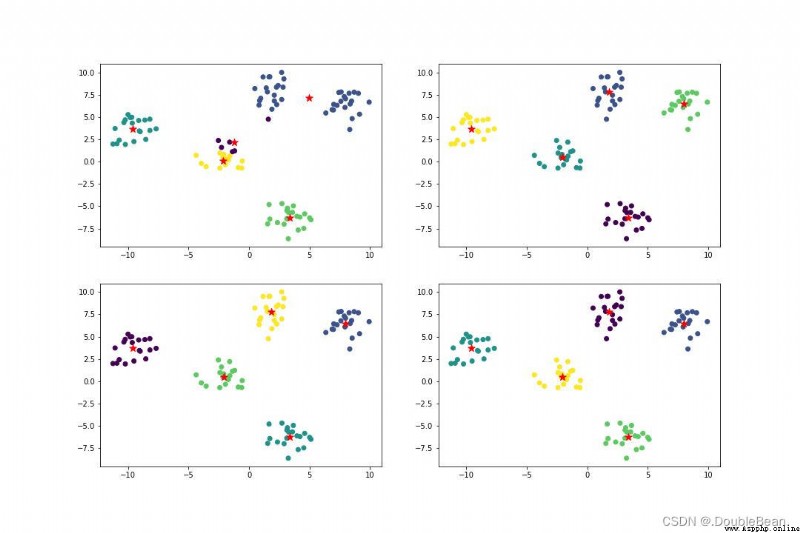
plt.show()
模型的穩定性提高了一些
SSE (sum of the squared errors,誤差平方和)
S S E = ∑ i = 1 k ∑ x j ∈ C i d i s t ( x j − μ i ) 2 SSE = \sum_{i=1}^{k} \sum_{x_j ∈ C_i} dist(x_j - μ_i)^2 SSE=i=1∑kxj∈Ci∑dist(xj−μi)2
其中,Ci是第i個類,xj是Ci中的樣本點,μi是Ci的類中心(Ci中所有樣本的均值),dist是歐幾裡得距離,SSE是所有樣本的聚類誤差,用來評估聚類效果的好壞,可以理解為損失函數
因為我們要考慮如何更好地更新類中心,使得對於所有樣本點,到其所屬類的類中心距離之和最小
m i n S S E = ∑ i = 1 k ∑ x j ∈ C i ( x j − μ i ) 2 = ∑ i = 1 k ∑ x j ∈ C i ( x j T x j − x j T μ i − μ i T x j + μ i T μ i ) = ∑ i = 1 k ( ∑ x j ∈ C i x j T x j − ( ∑ x j ∈ C i x j T ) μ i − μ i T ( ∑ x j ∈ C i x j ) + m i μ i T μ i ) \begin{aligned} min SSE =& \sum_{i=1}^{k} \sum_{x_j ∈ C_i} (x_j - μ_i)^2 \\ =& \sum_{i=1}^{k} \sum_{x_j ∈ C_i} (x_j^Tx_j - x_j^Tμ_i - μ_i^Tx_j + μ_i^Tμ_i) \\ =& \sum_{i=1}^{k} (\sum_{x_j ∈ C_i} x_j^Tx_j - (\sum_{x_j ∈ C_i} x_j^T)μ_i - μ_i^T(\sum_{x_j ∈ C_i} x_j) + m_i μ_i^Tμ_i) \end{aligned} minSSE===i=1∑kxj∈Ci∑(xj−μi)2i=1∑kxj∈Ci∑(xjTxj−xjTμi−μiTxj+μiTμi)i=1∑k(xj∈Ci∑xjTxj−(xj∈Ci∑xjT)μi−μiT(xj∈Ci∑xj)+miμiTμi)
其中,mi是第i個類中樣本點個數
對類中心求導:
∂ S S E ∂ μ i = − ( ∑ x j ∈ C i x j ) − ( ∑ x j ∈ C i x j ) + 2 m i μ i \frac{\partial SSE}{\partial μ_i} = - (\sum_{x_j ∈ C_i} x_j) - (\sum_{x_j ∈ C_i} x_j) + 2m_iμ_i ∂μi∂SSE=−(xj∈Ci∑xj)−(xj∈Ci∑xj)+2miμi
令 ∂ S S E ∂ μ i = 0 \frac{\partial SSE}{\partial μ_i} = 0 ∂μi∂SSE=0
μ i = 1 m i ∑ x j ∈ C i x j μ_i = \frac{1}{m_i}\sum_{x_j∈C_i} {x_j} μi=mi1xj∈Ci∑xj
由上式可知,類的最小化SSE的最佳類中心點是類中所有樣本點的均值
前面提到Kmeans的缺點中,有提到過k值的選取對K-means影響很大。引用的這個數據集可以很容易看出來數據應該劃分為5類,但不是所有數據集都能這麼容易得出k值的大小,一種選取k值的辦法就是通過不斷嘗試: 手肘法
手肘法的核心指標便是SSE
如圖所示便是該數據的肘部曲線圖,橫坐標是k值的選取,縱坐標是誤差平方和,曲線呈現遞降的趨勢,為什麼?
當k=1時,表示不對樣本點進行分類,直接認為所有樣本點為同一類。當 k = numSamples,即k為樣本點的個數,就是一個樣本點分為一類,每一個類的類中心就是樣本點自己本身,所以樣本點到自己的類中心的距離都為0,SSE就為0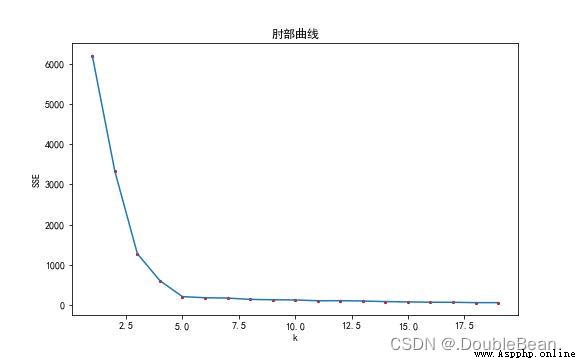
手肘法的核心思想是:隨著聚類數k的增大,樣本劃分會更加精細,每個簇的聚合程度會逐漸提高,那麼誤差平方和SSE自然會逐漸變小。並且,當k小於真實聚類數時,由於k的增大會大幅增加每個簇的聚合程度,故SSE的下降幅度會很大,而當k到達真實聚類數時,再增加k所得到的聚合程度回報會迅速變小,所以SSE的下降幅度會驟減,然後隨著k值的繼續增大而趨於平緩,也就是說SSE和k的關系圖是一個手肘的形狀,而這個肘部對應的k值就是數據的真實聚類數。當然,這也是該方法被稱為手肘法的原因
from pylab import *
mpl.rcParams['font.sans-serif'] = ['SimHei']
# 畫肘部法曲線
K = []
inertia = []
for k_num in range(20): # 這裡按理說應該是data.shape[0],但數據的樣本點太多,而且也沒必要
model = my_Kmeans_plus(k = k_num + 1, init = 'Kmeans++')
model.fit(data)
centers, ClusterAssment = model.predict(data)
K.append(k_num + 1)
inertia.append(np.sum(ClusterAssment[:, 1]))
plt.title('肘部曲線')
plt.xlabel('k')
plt.ylabel('SSE')
plt.plot(K, inertia)
plt.scatter(K, inertia, c='r', s=4)
plt.show()
調庫實現Kmeans並繪制手肘法
from sklearn.cluster import KMeans
from pylab import *
mpl.rcParams['font.sans-serif'] = ['SimHei']
sse = []
for k_num in range(1, 20):
model = KMeans(n_clusters=k_num, init="random", n_init=10, max_iter=200)
model.fit(data)
sse.append(model.inertia_)
plt.title('肘部曲線')
plt.xlabel('k')
plt.ylabel('SSE')
plt.plot(range(1, 20), sse)
plt.scatter(range(1, 20), sse, c='r', s=4)
plt.show()