Python腳本編輯
使用的文件
輸入 sys模塊
從命令行獲得文件名稱
進行序列信息統計的函數
使用def制作一個函數
.format使用:
進行函數計算
結果屏幕展示
結果輸出文件
腳本運行
Python腳本編輯使用Python對fasta格式的序列進行基本信息統計
預期設計輸出文件中包括fasta文件名,序列長度,GC含量以及ATCG各自的含量。
使用的文件輸入 sys模塊test.fasta
stat.py
#!/usr/bin/env pythonimport sys從命令行獲得文件名稱file_fasta = sys.argv[1]#獲得文件名file_name = file_fasta.split('.')name = file_name[0]sys.argv[1]模塊是從程序外部獲取參數的橋梁,可以將命令行的參數輸入到py程序內。
sys.argv[0]是程序本身,sys.argv[1]是程序後跟著第一個參數。
我們將文件名作為輸入參數,這一步在最後運行有展示。
在結束輸出時會輸出一個包含統計信息的txt文件,我們將用fasta文件名作為txt文件的前綴,所以我們需要獲取fasta文件的名字。
.split('.')是將file_fasta以.為分隔符分開會生成'test','txt',賦值給file_name則file_name會包含著兩個字符。
file_name[0]則是取第一個值'test',python中默認第一個數字是0,所以不能輸入1。
進行序列信息統計的函數序列的長度很好統計使用len函數即可,但是GC含量和ACTG的百分比計算需要費點事情。
使用def制作一個函數Python 使用def 開始函數定義,緊接著是函數名,括號內部為函數的參數,內部為函數的 具體功能實現代碼
def get_info(chr): chr = chr.upper() count_g = chr.count('G') count_c = chr.count('C') count_a = chr.count('A') count_t = chr.count('T')命名這個函數為get_info,內部參數為chr
在咱們會將fasta中ATCG的鹼基內容賦值給chr,鹼基可能有大寫有小寫,所以我們使用.upper將所以字符變成大寫。
再使用.count('G')統計ATCG各自的數量並賦值給對應count_g,我們用ATCG各自的統計數可以在後面計算中免疫N值干擾。
gc = (count_g + count_c) / (count_a + count_t + count_c + count_g)A = (count_a) / (count_a + count_t + count_c + count_g)T = (count_t) / (count_a + count_t + count_c + count_g)C = (count_c) / (count_a + count_t + count_c + count_g)G = (count_g) / (count_a + count_t + count_c + count_g)gc_con = '{:.2%}'.format(gc)A_content = '{:.2%}'.format(A)T_content = '{:.2%}'.format(T)C_content = '{:.2%}'.format(C)G_content = '{:.2%}'.format(G)return (gc_con,A_content,T_content,C_content,G_content)gc含量計算其等於(G的數量+C的數量)/(A的數量+T的數量+C的數量+G的數量)
A的含量等於(A的數量)/(A的數量+T的數量+C的數量+G的數量),其他值的計算以此類推。
.format使用:進行函數計算"{1} {0} {1}".format("hello", "world")設置指定位置。
'world hello world'
{:.2f} 保留小數點後兩位
最後,使用return返回函數結果(gc_con,A_content,T_content,C_content,G_content)
#進行函數計算with open(file_fasta,'r') as read_fa:讀取文件內容賦值給read_fa
python中有兩個方式打開文件一種是直接使用open("test.fasta","r"),執行完以後f.close()關閉。
注釋:"r"只讀模式打開文件;"w"以只寫模式打開文件,這種模式下輸入內容會覆蓋原有內容;"a"以追加模式打開一個文件,這個模式會把新內容追加到原有內容的末尾,不會覆蓋。
這裡使用的是第二方式with內置函數,它可以將文件自動關閉。
for val in read_fa: val = val.strip() if not val.startswith(">"): seq_info = get_info(val) len_fasta = len(val)將read_fa內容賦值給val。
strip() 方法用於移除字符串頭尾指定的字符(默認為空格或換行符),這裡使用默認。
然後使用startswith() 方法用於檢查字符串是否是以指定子字符串開頭,在當不是>開頭的行時候,才對核酸序列才進行信息統計。
len() 方法返回字符長度獲得片段長度
結果屏幕展示#結果屏幕展示print('******\n{0}\nlength:{1}\ngc content :{2}\nA content :{3}\nT content :{4}\nC content :{5}\nG content :{6}\n******'.format(name,len_fasta,seq_info[0],seq_info[1],seq_info[2],seq_info[3],seq_info[4]))使用\n進行換行,用.format指定值輸出位置。
結果輸出文件os.write(fd, str)
write() 方法用於寫入字符串到文件描述符 fd
#結果輸出文件file_output = open("{}sum.txt".format(name),'a')file_output.write('******\n')file_output.write('{}\n'.format(name))file_output.write('length:{:d}\n'.format(len_fasta))file_output.write('gc content :{}\n'.format(seq_info[0]))file_output.write('A content :{}\n'.format(seq_info[1]))file_output.write('T content :{}\n'.format(seq_info[2]))file_output.write('C content :{}\n'.format(seq_info[3]))file_output.write('G content :{}\n'.format(seq_info[4]))file_output.write('******')file_output.close()腳本運行執行腳本(linux系統)
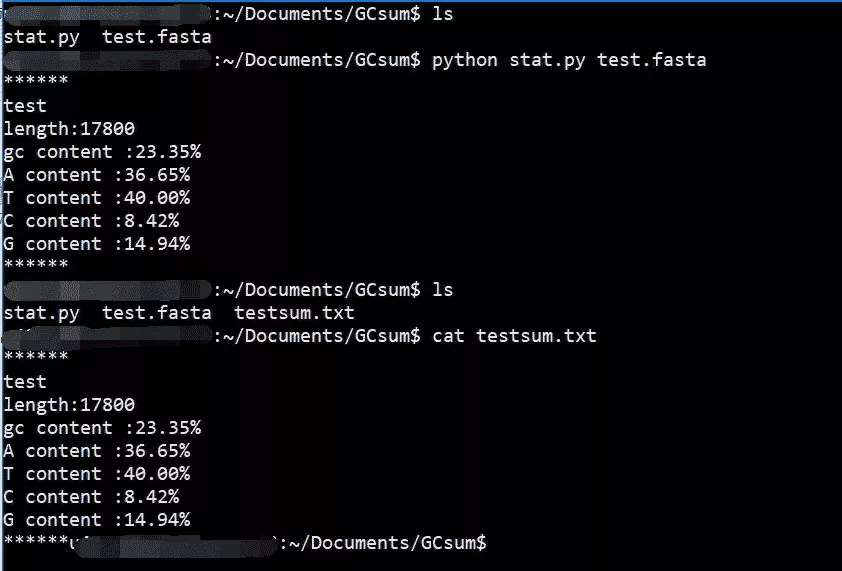
使用ls命令可以看到當前目錄下有已經寫好的py文件以及數據test.fasta。
運行時注意我們編寫時設置從命令行獲得文件名稱,所以要在後面跟上fasta文件,這樣才能成功運行。
運行結束後可以看見屏幕上有結果的打印,同時也生成了testsum.txt。
使用cat命令查看可以看到結果。
以上就是Python腳本提取fasta文件單序列信息實現的詳細內容,更多關於Python提取fasta單序列的資料請關注軟件開發網其它相關文章!