在解釋NII(NIFTI,neuroimaging information technology initiative)格式之前,我們需要先知道一下Analyze格式。
每一個Analyze格式的數據包含了兩個文件:帶有二進制圖像信息的.img,包含圖像元數據的頭文件.hdr
但是,Analyze的頭文件不能很真實地反應元數據(比如沒有方向信息,eg在左邊還是在右邊),於是NIFTI格式文件應運而生
nii格式(nifti格式的擴展)是為多維神經影像學發明的。一個nii格式主要包含三部分:hdr, ext, img
醫學影像數據有兩種格式,dicom和nii格式,他們定義了不同的方向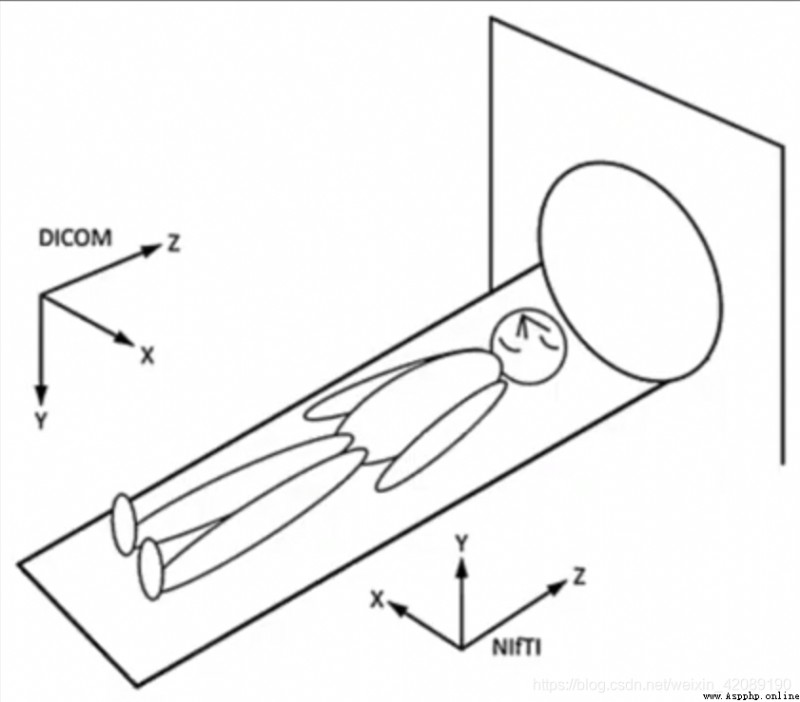
注意:使用這個軟件的時候,路徑中不能有中文
三視圖

import nibabel as nib
filename = 'CTC-1835078273_seg.nii'
img = nib.load(filename)
img.shape
#(512, 512, 539)一個nibable 圖像有三部分組合而成:
data = img.get_fdata()
data.shape,type(data)
#((512, 512, 539), numpy.memmap)至少需要一些image 數據 和一個image 坐標轉換矩陣(affine)
import numpy as np
data = np.random.randint((32, 32, 15, 100), dtype=np.int16)
img = nib.Nifti1Image(data, np.eye(4))
nib.save(img, 'exa.nii') 
這裡numpy.memmap是內存映像文件。它是一種將磁盤上的非常大的二進制數據文件當做內存中的數組進行處理的方式。它允許將大文件分成小段進行讀寫,而不是一次性將整個數組讀入內存。
memmap也擁有跟普通數組一樣的方法,因此,基本上只要是能用於ndarray的算法就也能用於memmap。
在官方文檔中,提供了一個人大腦的MRI圖像。
import nibabel as nib
epi_img = nib.load('someones_epi.nii.gz')
epi_img_data = epi_img.get_fdata()
epi_img_data.shape
#(53, 61, 33)[EPI數據]我們看看數組的第一維、第二維和第三維上的切片。
import matplotlib.pyplot as plt def show_slices(slices): """ Function to display row of image slices """ fig, axes = plt.subplots(1, len(slices)) for i, slice in enumerate(slices): axes[i].imshow(slice.T, cmap="gray", origin="lower") slice_0 = epi_img_data[0, :, :] slice_1 = epi_img_data[:, 0, :] slice_2 = epi_img_data[:, :, 0] show_slices([slice_0, slice_1, slice_2]) plt.suptitle("Center slices for EPI image")
image維度是(53, 61, 33),可以想成是53張61*33的圖,可以想成是61張53*33的圖,還可以想成是33張53*61的圖
[結構數據(解剖)]
anat_img = nib.load('someones_anatomy.nii.gz') anat_img_data = anat_img.get_fdata() print(anat_img_data.shape) #(57, 67, 56) show_slices([anat_img_data[0, :, :], anat_img_data[:, 0, :], anat_img_data[:, :, 0]]) plt.suptitle("Center slices for anatomical image")
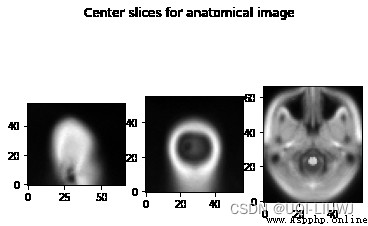
通常情況下,我們有不同的解剖掃描視野,因此解剖圖像具有不同的形狀、大小和方向。
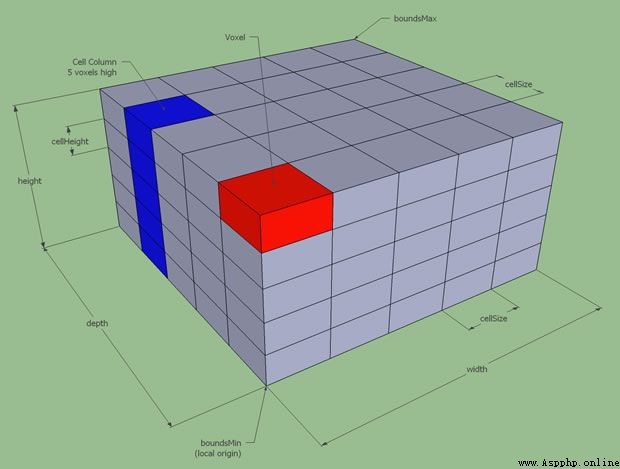
體素是具有體積的像素。
在上面的代碼中,來自 EPI 數據的 slice_0 是來自 3D 圖像的 2D 切片。 切片灰度圖像中的每個像素也代表一個體素,因為這個 2D 圖像代表了 3D 圖像中具有一定厚度的切片。
因此,3D 陣列也是體素陣列。 對於任何數組,我們都可以通過索引來選擇特定的值。 例如,我們可以像這樣獲取 EPI 數據數組中中間體素的值:
epi_img_data[0,0,0]
#10.755071640014648[0,0,0]就是體素坐標
體素坐標幾乎不告訴我們數據在掃描儀中的位置來自何處。 例如,假設我們有體素坐標 (26, 30, 16)。 如果沒有更多信息,我們不知道這個體素位置是在大腦的左側還是右側,還是來自掃描儀的左側或右側。 這是因為掃描儀允許我們在幾乎任何任意位置和方向收集體素數據。
比如我們對EPI圖,將掃描儀稍微旋轉一下,得到紅色框內的部分
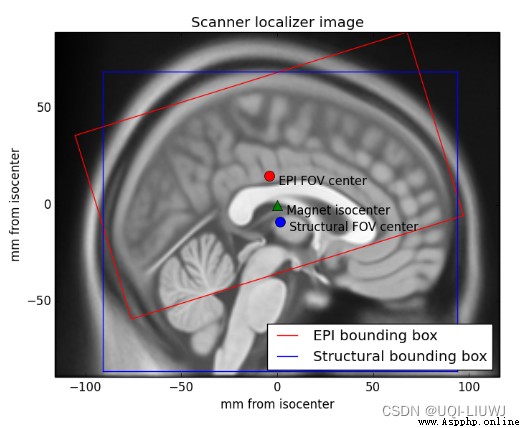
我們進行了解剖和 EPI 掃描,稍後我們肯定希望能夠將 somes_epi.nii.gz 中的數據與someones_anatomy.nii.gz 相關聯。目前我們不能輕易做到這一點,因為我們收集到的解剖圖像與 EPI 圖像具有不同的視野和方向。所以體素坐標暫時不能一一對應。
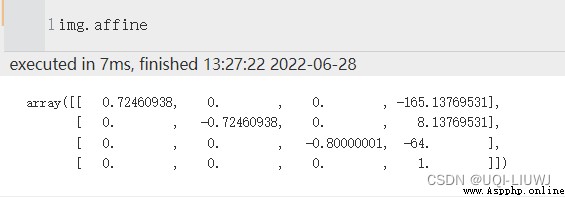
我們通過跟蹤體素坐標與某些參考空間的關系來解決這個問題。具體而言,仿射陣列(affine)存儲圖像數據中的體素坐標與參考空間中的坐標之間的關系
“參考空間”中的“空間”是什麼意思? 該空間由一組有序的軸定義。 對於我們的 3D 空間世界,它是一組 3 個獨立的軸。 我們可以通過選擇這些軸來決定我們想要使用的空間。 我們需要選擇軸的原點、方向和單位。(xyz-坐標軸,單位毫米)
我們希望將體素坐標(i,j,k)轉換到參考空間坐標(x,y,z),即(x,y,z)=f(i,j,k)
而基本的矩陣乘法,可以滿足
坐標軸伸縮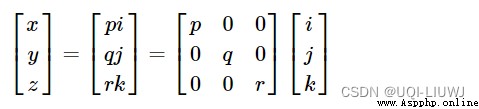 旋轉
旋轉圍繞第一個坐標軸旋轉
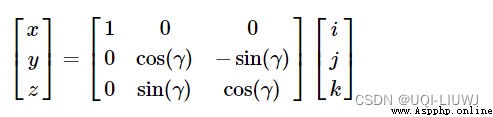
圍繞第二個坐標軸旋轉
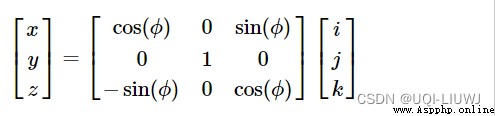
圍繞第三個坐標軸旋轉
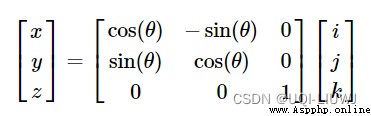
這幾個基本的操作相結合(矩陣乘法),就可以實現坐標軸的變換
上面完成了坐標軸的對應,但是體素的原點(0,0,0)對應的不一定是參考空間的(0,0,0)點,假設對應的是參考空間中的(a,b,c)坐標,那麼,體素中的(i,j,k)對應的是參考空間中的(x,y,z) 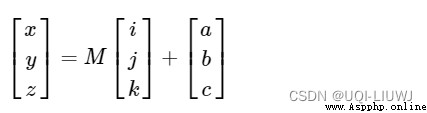
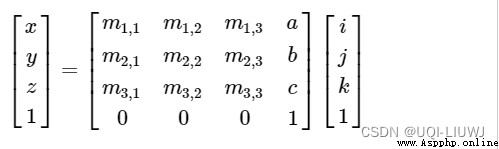
參考文獻
NII - Just Solve the File Format Problem (archiveteam.org)
How to read NIFTI format image (. nii file) (programmer.group)
什麼是體素(Voxel)? - 知乎 (zhihu.com)